DNA Sequenzierung
Die DNA-Sequenzierung stellt eine Standardmethode zur Identifizierung pathogener Genveränderungen (z. B. Punktmutationen, Deletionen/Duplikationen, Translokationen) in Tumoren dar. Dabei kommen am Institut verschiedene Techniken zum Einsatz.
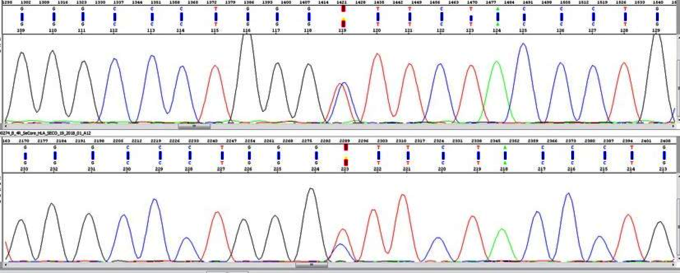
Die Sanger-Sequenzierung basiert auf dem Synthesekettenabbruch-Prinzip (Didesoxy-Methode). Über Kapillarelektrophorese und Laserdetektion wird der sukzessive Einbau der Desoxynucleotide (Adenin, Guanin, Cytosin, Thymin) während der enzymatischen DNA-Synthese bestimmt.
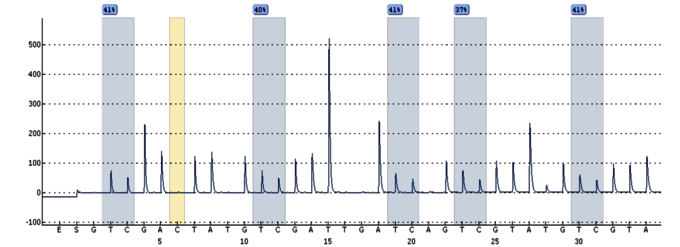
Die Pyrosequenzierung beruht auf der primerbasierten Synthese eines komplementären DNA-Stranges, wobei beim Einbau des korrekten Nukleotids ein Lichtsignal erzeugt wird. Dieses Signal wird in Echtzeit detektiert und mittels Auswerte-Software zu einer Nukleotidsequenz zusammengesetzt. Die Pyrosequenzierung ist besonders geeignet für die schnelle und hochauflösende Analyse kleiner genetischer und epigenetischer DNA-Aberrationen mit bekannter Genomposition.

Unter Next-Generation-Sequencing (NGS) versteht man eine Reihe innovativer Hochdurchsatztechnologien. Der Vorteil des NGS liegt in der massiven parallelen Sequenzierung von Millionen unterschiedlicher DNA-Genfragmente in einem einzigen Sequenzierlauf bei sehr hoher Sensitivität. NGS ist heute ein Standardverfahren in der molekularpathologischen Routinediagnostik der Onkologie. Am Institut werden mehrere, für bestimmte Tumorentitäten spezifische Genpanel (Amplikon- oder Hybrid capture-basiert) eingesetzt. Das NGS wird in einem partnerschaftlichen Labor mit ausgezeichneter Ausstattung und eigener Bioinformatik durchgeführt (https://pathonext.de).




